Què és l'array CGH?
És una tècnica emprada en biologia molecular per a la detecció de canvis a la dosi genòmica (delecions o duplicacions) amb un nivell de resolució molt superior al cariotip.
En què consisteix un array CHG?
Els arrays CGH (comparative genomic hybridization), també coneguts com a arrays d’hibridació genòmica comparada, permeten detectar canvis en la dosi genòmica (delecions o duplicacions) amb un nivell de resolució molt superior al cariotip.
Avantatges i desavantatges d'un array CGH
Avantatges d'un array CHG
Els avantatges d’aquesta tècnica són:
- Major sensibilitat i poder de resolució respecte al cariotip per detectar delecions, duplicacions o reordenaments desbalancejats.
- Possibilitat d’automatització.
- Menys temps de lliurament de resultats ja que no es requereix cultiu cel·lular per a l’obtenció de l’ADN.
Desavantatges d'un array CGH
Els desavantatges d’aquesta tècnica són que no pot detectar:
- Reordenaments cromosòmics equilibrats, com ara translocacions o inversions.
- La localització cromosòmica de les duplicacions (requereix una prova FISH addicional).
- Mosaïcisme de baixa freqüència (<30% de cèl·lules anormals).
- Alguns tipus de poliploïdia com la triploïdia.
- La disomia uniparental (UPD).
- Variacions en regions repetitives, com les expansions de triplet de la síndrome del cromosoma X fràgil.
- Defectes d’imprinting.
- Malalties genètiques causades per mutacions puntuals o herència multifactorial.
Aplicacions d'un array CHG
Aplicacions d'un array CHG prenatal
L’array CGH es fa servir en el diagnòstic prenatal:
- Per a una caracterització més acurada de les anomalies detectades per mètodes tradicionals (cariotip).
- Quan s’observen anomalies estructurals del fetus a l’ecografia o un retard del creixement.
- En els casos de mort fetal intrauterina o mortinats.
- A petició del pacient o del metge.
Els microarrays incrementen en un 5-10% la taxa de detecció d’alteracions cromosòmiques rellevants en embarassos d’alt risc i un 1-2% en els embarassos de risc baix. Com que de vegades aquesta tècnica (així com l’exoma i el genoma), detecten més variants de significat incert (VUS), és important que un professional expert interpreti les dades i assessori el metge i/o el pacient.
Aplicacions d'un array CHG postnatal
És la tècnica inicial d’elecció per a l’estudi de pacients amb:
- Retard del desenvolupament psicomotor/discapacitat intel·lectual
- Retard del creixement
- Trastorns de l’espectre autista
- Dificultats daprenentatge de moderades a greus
- Trets dismòrfics
- Amb malformacions congènites d’etiologia poc clara
És així perquè ofereixen un rendiment diagnòstic més gran (15-20%) comparat amb el cariotip. Aquesta darrera tècnica s’hauria d’utilitzar com a indicació primària per a l’estudi de pacients amb quadres cromosòmics reconeixibles, casos amb història familiar de reordenaments cromosòmics i en l’estudi de la infertilitat i/o avortaments múltiples.
Quina és la resolució d'un array CGH?
En comparació amb l’anàlisi cromosòmica convencional, el límit de detecció de la qual és de 5 a 10 Mb, l’array CGH aconsegueix detectar desequilibris genòmics amb una resolució diagnòstica de 50 a 100 kb.
Els arrays CGH es poden dissenyar per cobrir qualsevol regió d’interès i assolir diferents resolucions.
El nivell de resolució es determina considerant la mida de la sonda emprada, la distància genòmica entre les sondes o el nombre de sondes que té l’array. Què vol dir?
- Com més petit sigui el fragment de DNA utilitzat com a sonda i més a prop es trobin cada dues sondes consecutives, més gran la resolució.
- Un array de 60 k té 60.000 sondes o oligonucleòtids distribuïts al llarg del genoma.
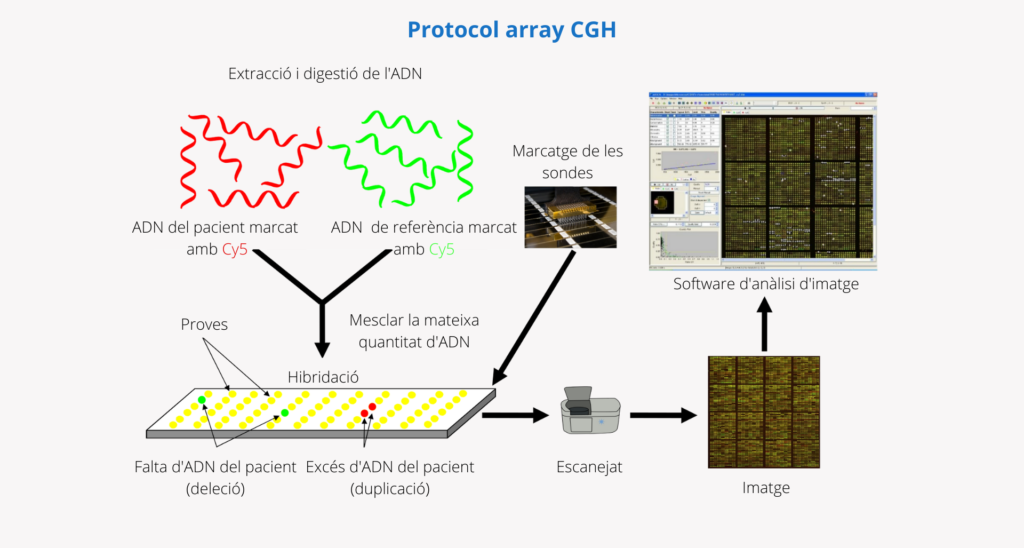
Com funciona un array CGH?
- En primer lloc, es procedeix a l’extracció i la digestió de l’ADN del pacient (a partir d’una mostra de sang, saliva, vellositats coriòniques, líquid amniòtic o altres cèl·lules o teixits).
- Després, la mateixa quantitat d’ADN del pacient i d’un ADN de referència (control) es marquen amb diferents fluoròfors.
- Posteriorment es barregen i es desnaturalitzen perquè passin de cadena doble a cadena senzilla.
- Al següent pas s’hibriden en un xip amb fragments d’ADN immobilitzats (sondes). Aquestes sondes cobreixen el genoma humà de la manera més uniforme possible i es poden personalitzar segons les necessitats de l’anàlisi.
- Els fragments d’ADN del pacient i d’ADN control competeixen per hibridar amb seqüències complementàries a les sondes de l’array.
- A continuació, es fa servir un escàner làser d’alta resolució per capturar i quantificar la intensitat de senyal fluorescent d’un color i l’altre que hibrida en cada sonda.
- Finalment, es calcula la relació de la intensitat de senyal fluorescent a l’ADN del pacient i l’ADN de referència per a cada sonda de l’array, obtenint-se informació sobre el nombre de còpies relatiu de seqüències al genoma al pacient en comparació amb el genoma de referència. Aquesta informació és interpretada per un programa d’anàlisi que permet assignar les dades a la regió cromosòmica respectiva i representar-les en forma de “cariotip molecular”.