¿Qué es el array CGH?
En una técnica empleada en biología molecular para la detección de cambios en la dosis genómica (deleciones o duplicaciones) con un nivel de resolución muy superior al cariotipo.
¿En qué consiste un array CHG?
Los arrays CGH (comparative genomic hybridization), también conocidos como arrays de hibridación genómica comparada, permiten detectar cambios en la dosis genómica (deleciones o duplicaciones) con un nivel de resolución muy superior al cariotipo.
Ventajas y desventajas de un array CGH
Ventajas de un array CHG
Las ventajas de esta técnica son:
- Mayor sensibilidad y poder de resolución respecto al cariotipo para detectar deleciones, duplicaciones o reordenamientos desbalanceados.
- Posibilidad de automatización.
- Menor tiempo de entrega de resultados ya que no se requiere cultivo celular para la obtención del DNA.
Desventajas de un array CGH
Las desventajas de esta técnica son que no puede detectar:
- Reordenamientos cromosómicos equilibrados, como translocaciones o inversiones.
- La localización cromosómica de las duplicaciones (requiere una prueba FISH adicional).
- Mosaicismo de baja frecuencia (<30% de células anormales).
- Algunos tipos de poliploidía como la triploidía.
- La disomía uniparental (UPD).
- Variaciones en regiones repetitivas, como las expansiones de triplete del síndrome del cromosoma X frágil.
- Defectos de impronta.
- Enfermedades genéticas causadas por mutaciones puntuales o herencia multifactorial.
Aplicaciones de un array CHG
Aplicaciones de un array CHG prenatal
El array CGH se utiliza en el diagnóstico prenatal:
- Para una caracterización más precisa de las anomalías detectadas por métodos tradicionales (cariotipo).
- Cuando se observan anomalías estructurales del feto en la ecografía o un retraso del crecimiento.
- En los casos de muerte fetal intrauterina o mortinatos.
- A petición del paciente o del médico.
Los microarrays incrementan en un 5-10% la tasa de detección de alteraciones cromosómicas relevantes en embarazos de alto riesgo y un 1-2% en los embarazos de bajo riesgo. Debido a que en ocasiones estas técnica (así como el exoma y genoma), detectan más variantes de significado incierto (VUS), es importante que un profesional experto interprete los datos y asesore al médico y/o al paciente.
Aplicaciones de un array CGH postnatal
Es la técnica inicial de elección para el estudio de pacientes con:
- Retraso del desarrollo psicomotor/discapacidad intelectual
- Retraso del crecimiento
- Trastornos del espectro autista
- Dificultades de aprendizaje de moderadas a graves
- Rasgos dismórficos
- Con malformaciones congénitas de etiología poco clara
Es así porque ofrecen un mayor rendimiento diagnóstico (15-20%) comparado con el cariotipo. Esta última técnica debería utilizarse como indicación primaria para el estudio de pacientes con cuadros cromosómicos reconocibles, casos con historia familiar de reordenamientos cromosómicos y en el estudio de la infertilidad y/o abortos múltiples.
¿Cuál es la resolución de un array CGH?
En comparación con el análisis cromosómico convencional, cuyo límite de detección es de 5 a 10 Mb, el array CGH consigue detectar desequilibrios genómicos con una resolución diagnóstica de 50 a 100 kb.
Los arrays CGH pueden diseñarse para cubrir cualquier región de interés y alcanzar distintas resoluciones.
El nivel de resolución se determina considerando el tamaño de la sonda empleada, la distancia genómica entre las sondas o el número de sondas que tiene el array. ¿Qué quiere decir?
- Cuanto más pequeño sea el fragmento de DNA utilizado como sonda y más cerca se encuentren cada dos sondas consecutivas, mayor la resolución.
- Un array de 60 k tiene 60.000 sondas u oligonucleótidos distribuidos a lo largo del genoma.
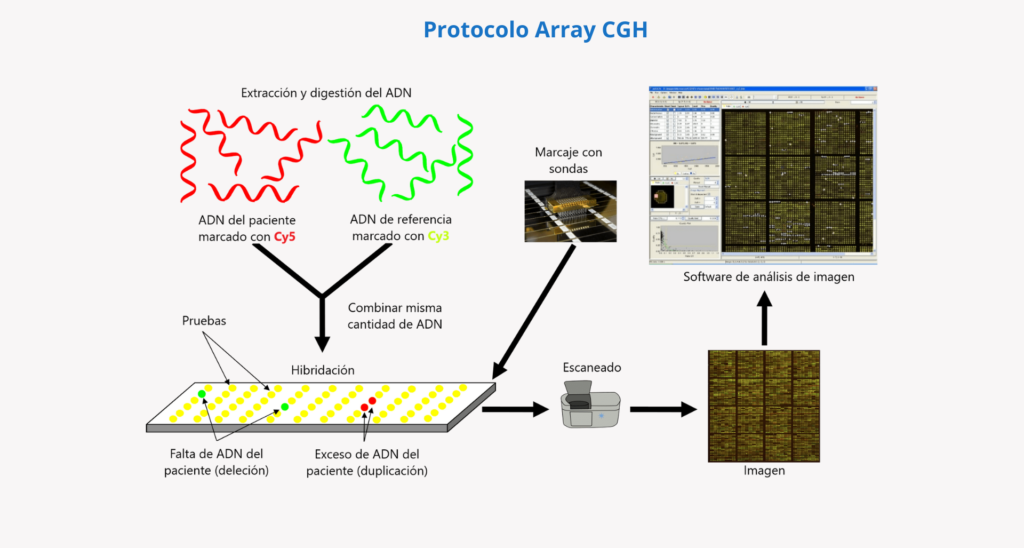
¿Cómo funciona un array CGH?
- En primer lugar, se procede a la extracción y digestión del ADN del paciente (a partir de una muestra de sangre, saliva, vellosidades coriónicas, líquido amniótico u otras células o tejidos).
- Tras ello, la misma cantidad de ADN del paciente y de un ADN de referencia (control) se marcan con diferentes fluoróforos.
- Posteriormente se mezclan y se desnaturalizan para que pasen de cadena doble a cadena sencilla.
- En el siguiente paso se hibridan en un chip con fragmentos de ADN inmovilizados (sondas). Estas sondas cubren el genoma humano de la manera más uniforme posible y pueden personalizarse según las necesidades del análisis.
- Los fragmentos de ADN del paciente y de ADN control compiten por hibridar con secuencias complementarias en las sondas del array.
- A continuación, se emplea un escáner láser de alta resolución para capturar y cuantificar la intensidad de señal fluorescente de uno y otro color que ha hibridado en cada sonda.
- Finalmente, se calcula la relación de la intensidad de señal fluorescente en el ADN del paciente y el ADN de referencia para cada sonda del array, obteniéndose información sobre el número de copias relativo de secuencias en el genoma en el paciente en comparación con el genoma de referencia. Esta información es interpretada por un software de análisis que permite asignar los datos a la región cromosómica respectiva y representarlos en forma de “cariotipo molecular”.