Was ist eine Array-CGH?
Ein in der Molekularbiologie verwendetes Verfahren zum Nachweis von genomischen Dosisveränderungen (Deletionen oder Duplikationen) mit einer viel höheren Auflösung als bei der Karyotypisierung.
Was ist eine Array-CHG?
Array-CGH (comparative genomic hybridisation), auch bekannt als vergleichende genomische Hybridisierungsarrays, ermöglichen den Nachweis von genomischen Dosisänderungen (Deletionen oder Duplikationen) mit einer Auflösung, die der Karyotypisierung weit überlegen ist.
Vor- und Nachteile einer Array-CGH
Vorteile einer Array-CGH
Die Vorteile dieser Technik sind:
- Höhere Empfindlichkeit und Auflösungsvermögen im Vergleich zur Karyotypisierung, um Deletionen, Duplikationen oder unbalancierte Rearrangements zu erkennen.
- Möglichkeit der Automatisierung.
- Kürzere Durchlaufzeit, da keine Zellkultur zur DNA-Gewinnung erforderlich ist.
Nachteile einer Array-CGH
Der Nachteil dieser Technik ist, dass sie folgendes nicht erkennen kann:
- Balancierte chromosomale Umlagerungen, wie Translokationen oder Inversionen.
- Chromosomale Lokalisierung von Duplikationen (erfordert einen zusätzlichen FISH-Test).
- Niederfrequenter Mosaizismus (<30% anormale Zellen).
- Einige Arten von Polyploidie wie Triploidie.
- Uniparentale Disomie (UPD).
- Variationen in sich wiederholenden Regionen, wie z. B. die Triplett-Erweiterungen beim fragilen X-Syndrom.
- Imprinting-Fehler.
- Genetische Krankheiten, die durch Punktmutationen oder multifaktorielle Vererbung verursacht werden.
Aplicaciones de un array CHG
Anwendungen einer pränatalen Array-CGH
Die Array-CGH wird in der Pränataldiagnostik eingesetzt:
- Für eine genauere Charakterisierung von Anomalien, die mit herkömmlichen Methoden (Karyotypisierung) festgestellt wurden.
- Wenn im Ultraschall strukturelle Anomalien des Fötus oder Wachstumsverzögerungen festgestellt werden.
- Bei intrauterinem Fötaltod oder Totgeburt.
- Auf Wunsch des Patienten oder des Arztes.
Microarrays erhöhen die Entdeckungsrate relevanter Chromosomenveränderungen bei Hochrisikoschwangerschaften um 5-10% und bei Niedrigrisikoschwangerschaften um 1-2%. Da diese Techniken (wie auch das Exom und das Genom) manchmal mehr Varianten unklarer Signifikanz (VUS) entdecken, ist es wichtig, dass ein Fachmann die Daten interpretiert und den Arzt und/oder den Patienten berät.
Anwendungen einer postnatalen Array-CGH
Es ist die erste Technik der Wahl für die Untersuchung von Patienten mit folgenden Problement:
- Psychomotorische Entwicklungsverzögerung/intellektuelle Behinderung
- Verzögertes Wachstum
- Autismus-Spektrum-Störungen
- Mäßige bis schwere Lernschwierigkeiten
- Dysmorphe Merkmale
- Angeborene Fehlbildungen unklarer Ätiologie
Dies liegt daran, dass sie im Vergleich zur Karyotypisierung eine höhere diagnostische Leistung (15-20%) bietet. Die Karyotipisierung sollte als primäre Indikation für die Untersuchung von Patienten mit erkennbaren Chromosomenmustern, von Fällen mit Chromosomenumlagerungen in der Familiengeschichte und bei der Untersuchung von Unfruchtbarkeit und/oder multiplen Fehlgeburten eingesetzt werden.
Wie hoch ist die Auflösung einer Array-CGH-Analyse?
Im Vergleich zur konventionellen Chromosomenanalyse, die eine Nachweisgrenze von 5 bis 10 Mb hat, kann die Array-CGH genomische Ungleichgewichte mit einer diagnostischen Auflösung von 50 bis 100 kb nachweisen.
CGH-Arrays können so konzipiert werden, dass sie jede beliebige Region von Interesse abdecken und unterschiedliche Auflösungen erreichen.
Der Grad der Auflösung wird durch die Größe der verwendeten Sonde, den genomischen Abstand zwischen den Sonden oder die Anzahl der Sonden im Array bestimmt. Was bedeutet das?
- Je kleiner das als Sonde verwendete DNA-Fragment ist und je näher zwei aufeinanderfolgende Sonden beieinander liegen, desto höher ist die Auflösung.
- Ein 60k-Array hat 60.000 Sonden oder Oligonukleotide, die über das gesamte Genom verteilt sind.
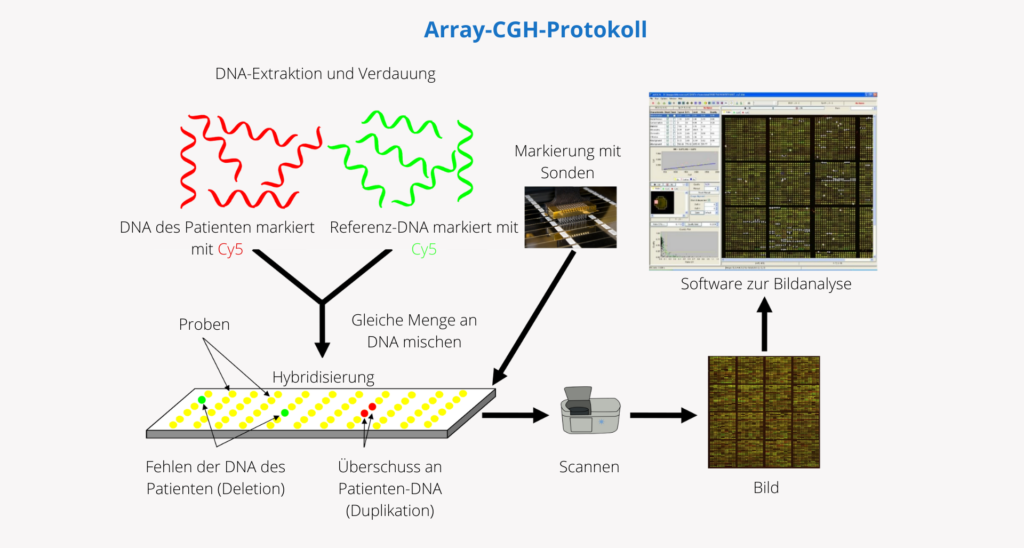
Wie funktioniert eine Array-CGH?
- Zunächst wird die DNA des Patienten extrahiert und verdaut (aus einer Blut-, Speichel-, Chorionzotten-, Fruchtwasser- oder einer anderen Zell- oder Gewebeprobe).
- Die gleiche Menge an Patienten-DNA und eine Referenz-DNA (Kontrolle) werden dann mit unterschiedlichen Fluorophoren markiert.
- Anschließend werden sie gemischt und von Doppelstrang auf Einzelstrang denaturiert.
- In einem nächsten Schritt werden sie auf einem Chip mit immobilisierten DNA-Fragmenten (Sonden) hybridisiert. Diese Sonden decken das menschliche Genom so gleichmäßig wie möglich ab und können je nach den Erfordernissen der Analyse individuell angepasst werden.
- Patienten- und Kontroll-DNA-Fragmente konkurrieren um die Hybridisierung mit komplementären Sequenzen auf den Array-Sonden.
- Mit einem hochauflösenden Laserscanner wird dann die Intensität des Fluoreszenzsignals jeder Farbe, die mit jeder Sonde hybridisiert hat, erfasst und quantifiziert.
- Schließlich wird das Verhältnis der Intensität des Fluoreszenzsignals in der DNA des Patienten und der Referenz-DNA für jede Sonde im Array berechnet, wodurch Informationen über die relative Kopienzahl der Sequenzen im Genom des Patienten im Vergleich zum Referenzgenom gewonnen werden. Diese Informationen werden von einer Analysesoftware interpretiert, die es ermöglicht, die Daten der jeweiligen chromosomalen Region zuzuordnen und als „molekularen Karyotyp“ darzustellen.