Was ist Exom-Sequenzierung?
Es handelt sich um eine Hochdursatz-Sequenzierungstechnik (NGS, next generation sequencing), die es ermöglicht, Millionen von DNA-Fragmenten parallel zu sequenzieren und alle Gene abzudecken (WES, whole exome sequencing).
Hochdurchsatz-Sequenzierung: Exom-Analyse
Die Sequenzierung des kompletten Genoms (WES) befasst sich mit (fast) allen Genen einer Person und ist in der Lage, Punktmutationen und andere Arten von genetischen Veränderungen (wie Insertionen und Deletionen) in der DNA zu identifizieren.
Wie bei der Genomsequenzierung wird auch hier der klinische Nutzen immer wichtiger. So berichten neuere Studien von einer Erfolgsquote bei der Diagnose seltener Krankheiten von knapp 25 %.
Vorteile und Nachteile der Ganz-Exom-Analyse
Vorteile der Ganz-Exom-Sequenzierung:
Die Vorteile dieser Technik sind:
- Sie bietet einen hochauflösenden, basenweisen Überblick über das Exom.
- Sie kann einzelne Nukleotidvarianten und Insertionen/Deletionen nachweisen.
- Schnellere Datenerfassung als bei der Genomanalyse. Im Allgemeinen sind „nur“ etwa 25.000 Varianten zu analysieren. Das heißt, es müssen weniger Daten gefiltert, untersucht und gespeichert werden.
- Derzeit befinden sich die meisten bekannten Punktmutationen, die mit Krankheiten in Verbindung gebracht werden, in der kodierenden Region (Exom) der DNA.
Nachteile der Ganz-Exom-Sequenzierung:
- Der Nachweis von CNVs (Kopienzahlveränderungen) ist nicht so zuverlässig wie die Exom-Sequenzierung.
- Es ist schwierig, große strukturelle Varianten zu erkennen.
- Erkennt keine Varianten in nicht kodierenden Regionen.
Klinische Anwendungen der Ganz-Exom-Analyse
Die Ganz-Exom-Analyse deckt mehrere Aspekte der Gesundheit ab:
- Identifizierung monogener Krankheiten (z. B. Thalassämie).
- Identifizierung der möglichen Ursache bei seltenen Krankheiten.
- Identifizierung einer Veranlagung zur Entwicklung einer polygenen Krankheit (z. B. Diabetes mellitus Typ 2).
- Identifizierung von Genträgern rezessiver Krankheiten, wie z. B. Mukoviszidose.
Die Bedeutung einer guten Datenauswertung
Die Exom-Sequenzierung ermöglicht die Identifizierung von viel mehr Varianten (Punktmutationen, Insertionen und Deletionen) als andere Techniken, obwohl die Bedeutung einiger dieser Informationen unbekannt ist. Da sich nicht alle genetischen Veränderungen auf die Gesundheit auswirken, ist es manchmal schwierig festzustellen, ob die identifizierten Varianten mit der Gesundheit der betreffenden Person oder der betreffenden Krankheit zusammenhängen. Manchmal wird eine identifizierte Variante mit einer anderen genetischen Störung in Verbindung gebracht, die noch nicht diagnostiziert wurde (sogenannte Zufalls- oder Sekundärbefunde).
Deshalb ist es wichtig, über ein Team von Experten zu verfügen, das in der Lage ist, die Daten zu interpretieren und die Fragen der Ärzte und Patienten zu beantworten.
Welche genetischen Varianten können mit einem Exom nachgewiesen werden?
Anwendungen der Ganz-Exom-Sequenzierung (WES, whole exome sequencing) sind der Nachweis von Einzelnukleotidveränderungen (SNV, single nucleotide variant oder SNP, single nucleotide polymorphism) oder Punktmutationen und kleinen Kopienzahlveränderungen wie kleinen Insertionen und Deletionen in kodierenden Regionen (Genen) der DNA.
Was besagt die Abdeckung?
Die Tiefe der Abdeckung (depth of coverage) gibt an, wie oft eine Genombasis sequenziert wurde. Je höher der Erfassungsgrad, desto zuverlässiger ist die Methode. Mit anderen Worten: Der Prozentsatz falsch negativer und falsch positiver Ergebnisse wird reduziert, und es ist sogar möglich, Varianten zu erkennen, die in Mosaik vorkommen.
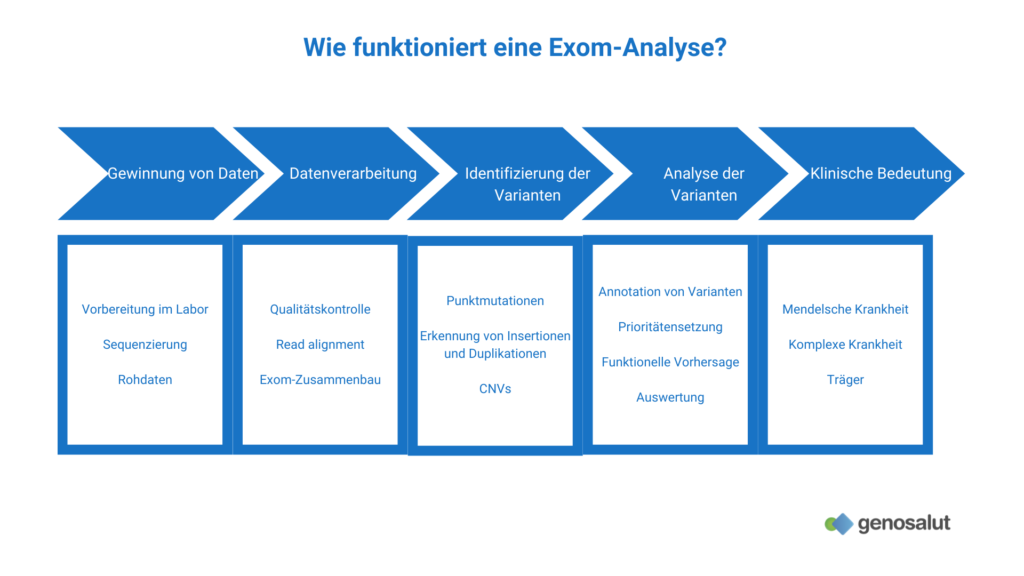
Wie funktioniert die Ganz-Exom-Sequenzierung?
Die Exom-Sequenzierung ist der Genom-Sequenzierung sehr ähnlich. Der Hauptunterschied besteht darin, dass ein Anreicherungsschritt der kodierenden Regionen (Gene) nach der Vorbereitung der Bibliotheken und vor der Amplifikation der Fragmente erforderlich ist.
Das heißt, das Exom muss vor der Sequenzierung vom Genom „getrennt“ werden, da es nur 1 % der gesamten Kern-DNA ausmacht. Der Grundgedanke der Anreicherung besteht darin, etwas von Interesse von anderen Stoffen zu trennen, indem man die Unterschiede in den physikalisch-chemischen Eigenschaften zwischen ihnen nutzt.
Es gibt zwei Hauptmethoden zur Anreicherung von Exomen:
- Array-basierte Exom-Anreicherung. Zur Erfassung des Exoms werden spezifische Sonden verwendet, die an hochdichten Mikroarrays befestigt sind. Nur diejenigen DNA-Fragmente, die eine komplementäre Sequenz besitzen, werden an das Microarray gebunden.
- Anreicherung des Exoms in Lösung mit biotinylierten Sonden. Auch hier werden nur die DNA-Fragmente mit einer komplementären Sequenz an die Sonden gebunden. Der Pool wird durch Zugabe von Streptavidin-Mikrosphären, die an die biotinylierten Sonden binden, in den gewünschten Regionen angereichert. Schließlich werden die mit den interessierenden Fragmenten angereicherten Streptavidin-Perlen magnetisch extrahiert.
